これから研究を始める高校生と指導教員のために 第2版
探究活動と課題研究の進め方・論文の書き方・口頭とポスター発表の仕方
(これ研)
副読文書
******* VIII-4. 一般化線形モデル入門 *******
説明変数が数値の場合の、目的変数の平均の依存性の解析
--- メダカにおける、体長と産卵数の関係 ---
(「これ研」の本文の第3部3.3.1, 3.3.2項;p. 93)

「これから研究を始める高校生と指導教員のために 第2版;探究活動と課題研究の進め方・論文の書き方・口頭とポスター発表の仕方」(これ研)の第3部6.6節「一般化線形モデルの奨め」(p. 133)で紹介している一般化線形モデルを解説します。本章では、説明変数が数値の場合の、目的変数の平均の依存性の解析の仕方を説明します。メダカにおける、体長と産卵数の関係の検定を例に用います(「これ研」の本文第3部3.3.1, 3.3.2項;p. 93)。「これ研」の第3部第6章「検定に挑戦しよう」(p. 118)を読み、検定とは何かを理解しておいて下さい。
RStudioを起動して下さい。起動方法の詳しい説明は、RStusioの起動の仕方を参照して下さい。そして、作業ディレクトリの指定の説明に従って作業ディレクトリを指定します。
setwd("/Users/sakai/Documents/書籍等原稿/これ研2版/課題研究解析")
# パソコン内での作業ディレクトリの位置がわかっている場合はこの方法が便利です。作業ディレクトリの位置の知り方は作業ディレクトリの表示を参照して下さい。あなたの作業ディレクトリの位置を""で囲んで書きます。この命令文を実行しておきます。
# 作業ディレクトリの位置がわからない(あるいは、上記の説明の意味がわからない)場合は、作業ディレクトリの指定の説明に従って指定して下さい。
作図にはtidyverseというものを用います。tidyverseをインストールしてありますか? まだならば、作図の準備の説明に従ってインストールして下さい。インストールしたら、tidyverseをRに読み込みます。
library(tidyverse)
# RStudioでこの命令文を実行しておきます。tidyverseを""で囲みません。Rstudio起動後、一度だけ実行すればよいです。Rstudioを終了して再び起動したときは再実行する必要があります。
解析では、データフレームの中の特定のデータ列を指定することを行います。指定の方法です。
データフレーム名$データ列名
# データフレーム名を書き、$を挟んで、指定したいデータ列名を書きます。
# 実行例
d$Angle.degree
d$Flying.distance.m
# データフレームdに入っているデータ列Angle.degree, flying.distance.mを指定します。
1. 解析に用いるcsvファイルのRへの読み込み
csvファイル「Medaka.csv」(画面1)に、メダカの地域(Locality)・体長(Length.mm)・産卵数(Egg.number)・体重(Weight.mg)が入っています(架空データです)。このcsvファイルのデータを使って解析と作図を行います。
画面1
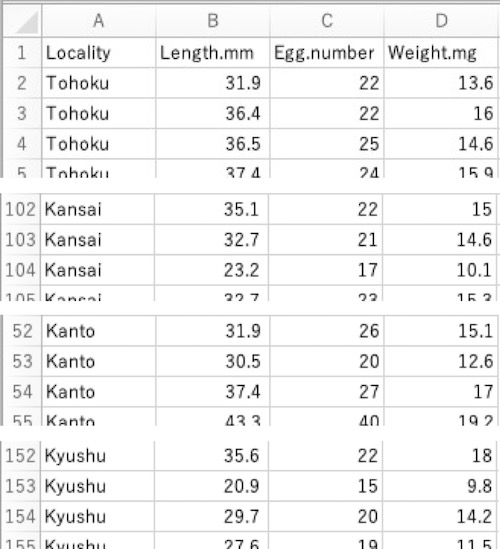
csvファイルをダウンロードして、ダウンロードしたファイルをあなたの作業ディレクトリに入れて下さい(「作業ディレクトリの指定」を参照)。
ご自身のデータを用いる場合は、そのデータが入ったcsvファイルを作業ディレクトリに入れて下さい。先頭行は必ずデータ名にして、続く行に個々のデータを書いて下さい。すべて、半角英数字で書いて下さい(日本語を入れない)。Excelで作ったファイルをcsvファイルに変換する方法は、Excelで作った解析用ファイルのcsv形式での保存を参照して下さい。
csvファイルをRに読み込み、データフレームに格納します。
d <- read.csv("Medaka.csv")
# csvファイル「Medaka.csv」を読み込んでデータフレームdに格納します。ファイル名を""で囲みます。ファイル名の拡張子「.csv」も忘れずに書きます。
# データフレームの名称(この例ではd)はお好みのものでよいです。
以下では、東北のメダカを対象に解析する場合を説明します。そのためにまず、東北のデータを取り出します。他の地域を解析する場合はその地域のデータを取り出して下さい。
d2 <- filter(d, Locality == "Tohoku")
# データフレームdのデータ列Localityに地域が入っています。Tohokuのメダカのデータを取りだしてデータフレームd2に格納します。=を2つ繋げ==とすることに注意して下さい。地域名は文字情報なので、地域名を""で囲みます。
地域を区別せずに全地域のデータをまとめて解析してしまう場合はデータの取り出しは不要です。もともと一つの母集団のデータしか入っていないファイルを解析する場合も取り出し不要です。
2. 一般化線形モデルによる解析
一般化線形モデル実行の手順に従って一般化線形モデルを実行します。
2.1. 適用する確率分布の指定
適用する確率分布を指定します。確率分布とはと確率分布の説明を読んで下さい。下の説明に従い、産卵数のヒストグラムを描いてみましょう。
画面2
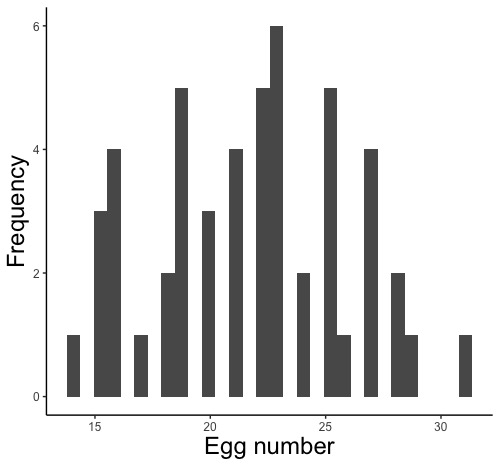
左右対称の釣り鐘方に近いので、確率分布を正規分布にします。
# 命令文が続く場合には+で繋げ、最後の命令文の後には+を付けません。
# 必須命令文
ggplot(d2, aes(x = Egg.number)) +
# データフレームd2を指定します。
# x = Egg.numberとして、ヒストグラムを描く体長のデータ列Egg.numberをx軸(横軸)に指定します。xは小文字です。
# 末尾の+を忘れないで下さい。
# ヒストグラムにおける、作図に用いるデータフレームとデータの指定
geom_histogram() +
# 描く図としてヒストグラムを指定します。
# 末尾の+を忘れないで下さい。ただし、以降の命令文を省略する場合は+を付けず、「geom_histogram()」とします。
# 省略してもよい命令文
# 以降の命令文を省略するとデフォルトで自動で描きます。
labs(x = "Egg number", y = "Frequency") +
# x軸(横軸)の名称をEgg numberに、y軸(縦軸)の名称をFrequencyにします。名称を""で囲みます。x, yは小文字です。
scale_y_continuous(limits = c(0, 6)) +
# y軸(縦軸)を描く範囲を0から6に指定します。ヒストグラムの場合は、y軸の最小値を必ず0にします。
theme_classic() +
# 図の背景色を白にします。
theme(
axis.title.x = element_text(size = 18),
axis.text.x = element_text(size = 9),
axis.title.y = element_text(size = 18),
axis.text.y = element_text(size = 9)
)
# x軸(横軸)・y軸(縦軸)の名称(axis.title.x, axis.title.y)の文字の大きさを18に、x軸・y軸の目盛り(axis.text.x, axis.text.y)の文字の大きさを9に指定します。
ヒストグラムの描き方の詳しい説明はメダカの体長のヒストグラムを参照して下さい。図の軸の説明の文字を日本語にしたい場合は、その方法も、メダカの体長のヒストグラムを参照して下さい。
2.2. 適用する連結関数の指定
適用する連結関数を指定します。説明変数の値と応答変数の平均値の関係性:連結関数を用いた解析と適用する連結関数の指定の説明を読んで下さい。下の説明に従い、メダカの体長と産卵数の散布図を描いてみましょう。
画面3
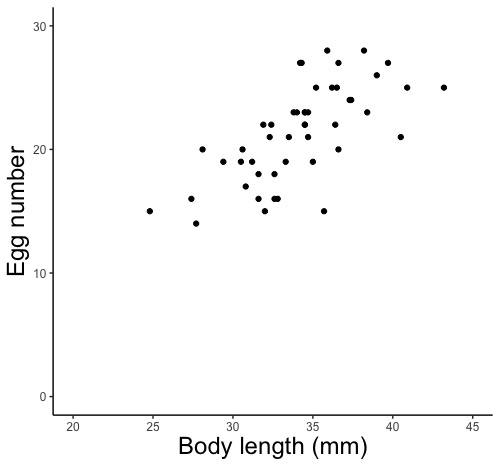
比例関係をしているようです。連結関数を比例(identity)にします。
# 命令文が続く場合には+で繋げ、最後の命令文の後には+を付けません。
# 必須命令文
ggplot(d2, aes(x = Length.mm, y = Egg.number)) +# データフレームd2を指定します。
# x = Length.mmとして、体長(Length.mm)が入っているデータ列Length.mmをx軸(横軸)に指定します。xは小文字です。
# y = Egg.numberとして、産卵数(Egg.number)が入っているデータ列Egg.numberをy軸(縦軸)に指定します。yは小文字です。
# 末尾の+を忘れないで下さい。
# 散布図における、作図に用いるデータフレームとデータの指定
geom_point() +
# 描く図として散布図を指定します。
# 末尾の+を忘れないで下さい。ただし、以降の命令文を省略する場合は+を付けず、「geom_point()」とします
# 省略してもよい命令文
# 以降の命令文を省略するとデフォルトで自動で描きます。
labs(x = "Body length (mm)", y = "Egg number") +# x軸(横軸)の名称をBody length (mm)に、y軸(縦軸)の名称をEgg numberにします。名称を""で囲みます。x, yは小文字です。
scale_x_continuous(limits = c(20, 45)) +
# x軸(横軸)を描く範囲を20から45に指定します。
scale_y_continuous(limits = c(0, 30)) +
# y軸(縦軸)を描く範囲を0から30に指定します。
theme_classic() +
# 図の背景色を白にします。
theme(
axis.title.x = element_text(size = 18),
axis.text.x = element_text(size = 9),
axis.title.y = element_text(size = 18),
axis.text.y = element_text(size = 9)
)
# x軸(横軸)・y軸(縦軸)の名称(axis.title.x, axis.title.y)の文字の大きさを18に、x軸・y軸の目盛り(axis.text.x, axis.text.y)の文字の大きさを9に指定します。
散布図の描き方の詳しい説明はメダカにおける、体長と産卵数の関係を参照して下さい。図の軸の説明の文字を日本語にしたい場合は、その方法も、メダカにおける、体長と産卵数の関係を参照して下さい。
2.3. 一般化線形モデルの実行
一般化線形モデルの命令文を実行します。一般化線形モデルの命令文の実行の説明を読んで下さい。説明変数が数値なので、1. 一般化線形モデルの命令文のみを実行します。
r <- glm(formula = Egg.number ~ Length.mm, family = gaussian(link = identity), data = d2)
summary(r)
# 一般化線形モデルの命令文を実行して、その結果をデータフレームrに格納します。
# 解析するデータフレーム;d2
# 応答変数;産卵数(Egg.number)
# 説明変数;体長(Length.mm)
# 確率分布;正規分布(gaussian)
# 連結関数;比例(identity)
# summary(r)を実行して、解析結果rの詳細を表示させます。
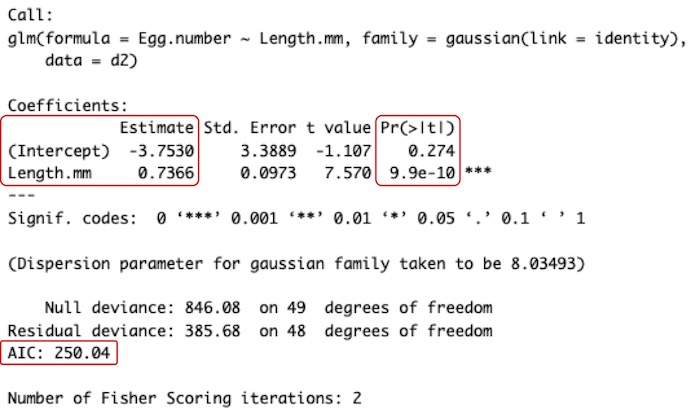
# 左上の赤枠内にある(Intercept)が、回帰式y = ax + bの切片bの値です。Length.mmが傾きaの値です。
# 右の赤枠内にあるのがP値です。切片(Intercept)の値(bの値)および傾きLength.mmの値(aの値)が有意に0と異なるのかどうかを示しています。切片b(Intercept)のP値は0.274なので、有意に0と異なるとはいえません。「e-」とあるのは10のマイナス何乗かということです。「9.9e-10」は「9.9 × 10- 10」です。傾きLength.mm(aの値)のP値は9.9e-10なので、有意に0と異なります。Length.mm(傾きaの値)は0.7366という正の値ですので、正の依存性があるということです。つまり、体長が大きいメダカほど産卵数が多いということです。
# 下の赤枠内の数値がAICです。AICによる、確率分布と連結関数の選択を参照して下さい。この値が一番小さい、確率分布と連結関数の組合せを採用します。他の確率分布(ガンマ分布・ポアソン分布)や他の連結関数(指数・S字・反比例)の組合せを色々と試すと、AICは、250.04よりも大きくなります。正規分布と比例の連結関数の組合せが良いということです。
3. 散布図への回帰式の描き込み
傾きが0から有意に異なっていたら、一般化線形モデルで得た回帰式を散布図に描き込みます(切片が0から有意に異なっていなかったとしても描き込みます)。メダカにおける体長と産卵数の作図でも回帰式を描きましたが、これは、y = ax + bという式(比例の式)のみに対応したものです。一般化線形モデルは、比例のみならず、指数・S字・反比例などの回帰式にも対応しています(説明変数の値と応答変数の平均値の関係性:連結関数を用いた解析参照)。こうした色々な回帰式を描き込む方法を紹介します。
傾きが0から有意に異なっていなかった場合は回帰式を描きません。切片が0から有意に異なっていたとしても、傾きが有意でなかったら回帰式は不要です。
画面4
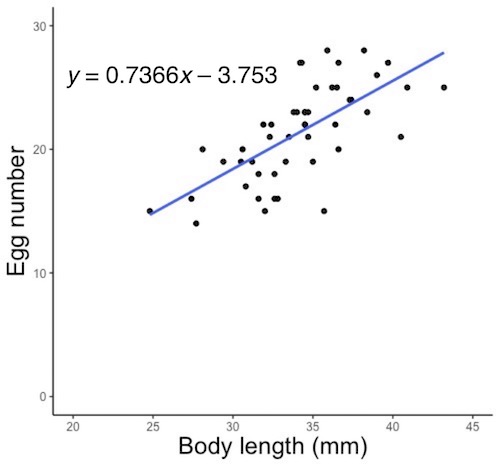
下記の方法でRで作図したら、それをPowerPoint等に読み込んで回帰式の値(この例ではy = 0.7366x - 3.753)を書き込みます。読み込む方法は図の保存・コピーを参照して下さい。
# 命令文が続く場合には+で繋げ、最後の命令文の後には+を付けません。
# 必須命令文
ggplot(d2, aes(x = Length.mm, y = Egg.number)) +# データフレームd2を指定します。
# x = Length.mmとして、体長(Length.mm)が入っているデータ列Length.mmをx軸(横軸)に指定します。xは小文字です。
# y = Egg.numberとして、産卵数(Egg.number)が入っているデータ列Egg.numberをy軸(縦軸)に指定します。yは小文字です。
# 末尾の+を忘れないで下さい。
# 散布図における、作図に用いるデータフレームとデータの指定
geom_point() +
# 描く図として散布図を指定します。
# 末尾の+を忘れないで下さい。
stat_smooth(method = glm, method.args = list(family = gaussian(link = identity)), se = FALSE) +
# 一般化線形モデルで得た回帰式を描き込みます。
# familiy = と link = で、解析で用いた確率分布(gaussian)と連結関数(identity)を指定します。必ず、一般化線形モデルの解析で指定したものと同じものを指定して下さい。そうでないと、解析で得られた回帰式とは異なる回帰式を描いてしまいます。
# link = で指定した連結関数に応じて、比例・指数・S字・反比例などの回帰式を自動的に描きわけます。
# se = FALSEは、信頼区間というものを描かせないという指定です。高校のレベルを超えているので描かせません。信頼区間の意味を理解しているのなら描かせてもよいです。その場合は、se = FALSEを取り除きます。
# 傾きが0から有意に異なっていなかった場合は回帰式を描きません。この命令文を省略します。
# 末尾の+を忘れないで下さい。ただし、以降の命令文を省略する場合は+を付けず、「stat_smooth(method = glm, method.args = list(family = gaussian(link = identity)), se = FALSE)」とします。
# 省略してもよい命令文
# 以降の命令文を省略するとデフォルトで自動で描きます。
labs(x = "Body length (mm)", y = "Egg number") +# x軸(横軸)の名称をBody length (mm)に、y軸(縦軸)の名称をEgg numberにします。名称を""で囲みます。x, yは小文字です。
scale_x_continuous(limits = c(20, 45)) +
# x軸(横軸)を描く範囲を20から45に指定します。
scale_y_continuous(limits = c(0, 30)) +
# y軸(縦軸)を描く範囲を0から30に指定します。
theme_classic() +
# 図の背景色を白にします。
theme(
axis.title.x = element_text(size = 18),
axis.text.x = element_text(size = 9),
axis.title.y = element_text(size = 18),
axis.text.y = element_text(size = 9)
)
# x軸(横軸)・y軸(縦軸)の名称(axis.title.x, axis.title.y)の文字の大きさを18に、x軸・y軸の目盛り(axis.text.x, axis.text.y)の文字の大きさを9に指定します。
散布図の描き方の詳しい説明はメダカにおける、体長と産卵数の関係を参照して下さい。図の軸の説明の文字を日本語にしたい場合は、その方法も、メダカにおける、体長と産卵数の関係を参照して下さい。